Distribución de frecuencias del polimorfismo T1236C en el gen ABCB1 en las poblaciones peruanas y comparación con poblaciones de América, Asia, Europa y África
DOI:
https://doi.org/10.24265/horizmed.2025.v25n3.07Palavras-chave:
Genes, Proteína ABCB1 , Poblaciones, Altitud , Perú , SaludResumo
Objetivo: Las mutaciones en el gen ABCB1 tienen un impacto en la respuesta a diversos fármacos
y aparentemente estarían también asociados a la génesis de neoplasias. El presente estudio explora y compara la distribución de frecuencias de la variación genética 1236T>C en el gen ABCB1 en nueve poblaciones peruanas de diferentes procedencias geográficas y ubicadas en diferentes niveles de altitud, así como las frecuencias genotípicas y alélicas reportadas en diferentes continentes. Materiales y métodos: El muestreo se llevó a cabo en los siguientes departamentos y localidades: Lima (n = 8), Loreto-Andoas (n = 16), San Martín-Lamas (n = 16),
Puno-Uros (n = 7), Puno-Taquile (n = 5), Arequipa-Chivay (n = 10), Arequipa-Cabanaconde (n = 6),
Ancash-Parobamba (n = 10) y Apurímac-Andahuaylas (n = 10). Se recolectó la muestra biológica, sangre periférica y/o células epiteliales por hisopado bucal. La extracción de ADN se efectuó mediante una técnica estándar. La determinación del polimorfismo 1236T>C (rs1128503) se realizó mediante reacción en cadena de la polimerasa (PCR) en tiempo real. Asimismo, se consideraron variables relacionadas con la procedencia geográfica (costa, sierra o selva), y la altitud (<2500 m s. n. m. o >2500 m s. n. m.). Para los análisis estadísticos, se aplicaron la prueba de
equilibrio de Hardy-Weinberg y la prueba X2 de Pearson, y se consideró un nivel de significancia
de valor p <0,05 y un intervalo de confianza (IC) al 95 %. Resultados: Se determinó la distribución
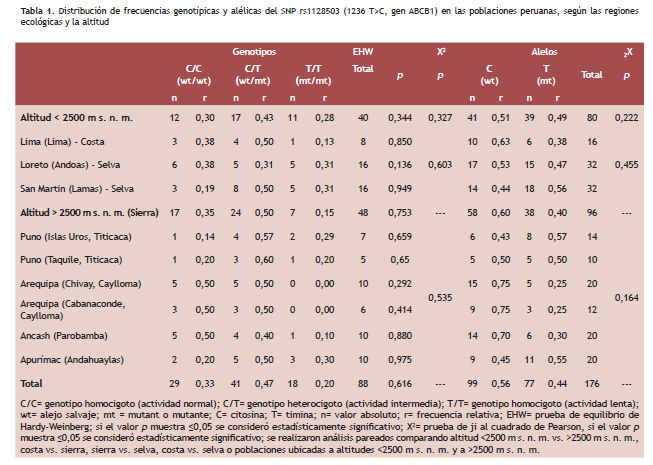
de frecuencias de la variación 1236T>C en el gen ABCB1 en las poblaciones peruanas. Según su
distribución geográfica en la costa, sierra y selva, en general, se observa una predominancia del
genotipo heterocigoto C/T (asociado con una actividad intermedia de la glicoproteína-P), con
la frecuencia más alta (60 %) registrada en la isla Taquile (Lago Titicaca, Puno). Considerando a la población peruana en conjunto (n = 88), la frecuencia fue de 43 % en localidades ubicadas a altitudes <2500 m s. n. m. y de 50 % a altitudes ≥2500 m s. n. m. En contraste, los homocigotos (C/C – actividad normal y T/T – actividad lenta) mostraron frecuencias similares (30 % y 28 %, respectivamente) a altitudes <2500 m s. n. m., pero distintas (35 % y 15 %, respectivamente)
a altitudes >2500 m s. n. m. En suma, a nivel de país, la distribución de los genotipos muestra lo siguiente: 47 % de C/T, 33 % de C/C y 20 % de T/T. Por otra parte, la prueba de equilibrio de Hardy-Weinberg no mostró ninguna significancia estadística. Conclusiones: El polimorfismo 1236 T>C en el gen ABCB1 en la población peruana presenta una predominancia del genotipo heterocigoto C/T, según la región geográfica y la altitud. A nivel de países o continentes, en general, se observa esa misma tendencia.
Downloads
Referências
Bukowski K, Kciuk M, Kontek R. Mechanisms of multidrug resistance in
cancer chemotherapy. Int J Mol Sci. 2020;21(9):3233. Disponible en:
https://doi.org/10.3390/ijms21093233
Elmeliegy M, Vourvahis M, Guo C, Wang DD. Effect of p-glycoprotein
(P-gp) inducers on exposure of p-gp substrates: review of clinical
drug–drug interaction studies. Clin Pharmacokinet. 2020;59(6):699–
Disponible en: https://doi.org/10.1007/s40262-020-00867-1
Tian Y, Lei Y, Wang Y, Lai J, Wang J, Xia F. Mechanism of multidrug
resistance to chemotherapy mediated by P‑glycoprotein (Review).
Int J Oncol. 2023;63(5):119. Disponible en: https://doi.org/10.3892/
ijo.2023.5567
Patel D, Sethi N, Patel P, Shah S, Patel K. Exploring the potential of
P-glycoprotein inhibitors in the targeted delivery of anti-cancer drugs:
A comprehensive review. Eur J Pharm Biopharm. 2024;198:114267.
Disponible en: http://dx.doi.org/10.1016/j.ejpb.2024.114267
Seelig A. P-Glycoprotein: one mechanism, many tasks and the consequences
for pharmacotherapy of cancers. Front Oncol. 2020;10:576559. Disponible
en: https://doi.org/10.3389/ fonc.2020.576559
Murakami T, Bodor E, Bodor N. Factors and dosage formulations
affecting the solubility and bioavailability of P-glycoprotein substrate
drugs. Expert Opin Drug Metab Toxicol. 2021;17(5):555-580. Disponible
en: https://doi.org/10.1080/17425255.2021.1902986
Hernández-Lozano I, Mairinger S, Filip T, Sauberer M, Wanek T, Stanek
J, et al. PET imaging to assess the impact of P-glycoprotein on
pulmonary drug delivery in rats. J Control Release. 2022;342:44-52.
Disponible en: https://doi.org/10.1016/j.jconrel.2021.12.031.
Ahmed Juvale II, Abdul Hamid AA, Abd Halim KB, Che Has AT.
P-glycoprotein: new insights into structure, physiological function,
regulation and alterations in disease. Heliyon. 2022;8(6):e09777.
Disponible en: https://doi.org/10.1016/j.heliyon.2022.e09777
Hodges LM, Markova SM, Chinn LW, Gow JM, Kroetz DL, Klein TE,
et al. Very important pharmacogene summary: ABCB1 (MDR1,
P-glycoprotein). Pharmacogenet Genomics. 2011;21(3):152-61.
Disponible en: https://doi.org/10.1097/FPC.0b013e3283385a1c
PharmGKB. ABCB1 [Internet]. Stanford: PharmGKB; 2025. Disponible
en: https://www.pharmgkb.org/gene/PA267/clinicalAnnotation
OMIM. Online Mendelian Inheritance in Man [Internet]. Baltimore: Johns
Hopkins University; 2025. Disponible en: https://www.omim.org/
Valencia E, Marcos P, Barbosa E, Sandoval J, Salazar A. Geographic
distribution of the 3435C>T polymorphism of the MDR1 gene in
Peruvian populations. Drug Metab Pers Ther. 2019;34(3). Disponible
en: https://doi.org/10.1515/dmpt-2018-0041
Salazar A, Youn-Ho K, Figueroa-Tataje J, Quijano F, Ore-Chávez D,
Sandoval-Sandoval J. Frecuencia del polimorfismo 282 C>T del gen
N-Acetiltransferasa (NAT2) en poblaciones peruanas e implicancias
en la salud. Horiz Med. 2016;16(1):20-31. Disponible en: https://doi.
org/10.24265/horizmed.2016.v16n1.04
Salazar A, Sandoval J, Mendizábal R, Kikushima F, Madsen H, Garred P,
et al. Variantes del gen Mannose Binding Lectin (MBL) en pobladores
amazónicos de Andoas-Loreto y su posible implicancia en la salud.
Horiz Med. 2006;6(1):13-9. Disponible en: https://doi.org/10.24265/
horizmed.2006.v6n1.02
Valencia E, Chevarría M, Coelho E, Sandoval J, Salazar A. Metabolizer
phenotype prediction in different Peruvian ethnic groups through
CYP2C9 polymorphisms. Drug Metab Pers Ther. 2021;36(2):113-21.
Disponible en: https://doi.org/10.1515/dmpt-2020-0146
Alvarado-Yarasca A, Sullón L, Salazar A, Loja B, Miyasato J,
Li-Amenero C, et al. Estudio de las variantes alélicas del gen CYP2C9
y monitorización clínica del valproato en plasma como fundamento de
la Medicina Personalizada. Diagnóstico. 2018;57(2):73-8. Disponible
en: https://doi.org/10.33734/diagnostico.v57i2.79
Marcos-Carbajal P, Allca-Muñoz C, Barbosa-Coelho E, Sandoval-
Sandoval J, Salazar-Granara A. Distribución por procedencia y altitud
del polimorfismo RS5522 del gen NR3C2 receptor de mineralocorticoide
en poblaciones peruanas. Rev Cuerpo Med HNAAA. 2024;17(2).
Disponible en: https://doi.org/10.35434/rcmhnaaa.2024.172.2198
Alvarado AT, Salazar-Granara A, Varela N, Quiñones LA, Li-Amenero C,
Bendezú MR, et al. Prevalence of GSTM1*0 and CYP1A1*2A (rs4646903)
variants in the central Peruvian coastal population: Pilot Study of
predictive genetic biomarkers for 4P medicine. Pharmacia. 2025;72:1–
Disponible en: https://doi.org/10.3897/pharmacia.72.e145034
Alvarado AT, Saravia M, Losno R, Pariona R, María Muñoz A,
Ybañez-Julca RO, et al. CYP2D6 and CYP2C19 genes associated with tricontinental and Latin American ancestry of Peruvians
;16(1):14–26. Disponible en: https://doi.org/10.2174/18723128
Alvarado AT, Ybañez-Julca R, Muñoz AM, Tejada-Bechi C, Cerro R,
Quiñones LA, et al. Frequency of CYP2D6*3 and *4 and metabolizer
phenotypes in three mestizo Peruvian populations. Pharmacia
;68(4): 891-898. Disponible en: https://doi.org/10.3897/
pharmacia.68.e75165
Sandoval JR, Salazar-Granara A, Acosta O, et al. Tracing the genomic
ancestry of Peruvians reveals a major legacy of pre-Columbian
ancestors. J Hum Genet. 2013;58(9):627–634. Disponible en: https://
doi.org/10.1038/jhg.2013.73
Harrison P, Ridwan A, Austine-Orimoloye O, Azov A, Barba M, Barnes I,
et al. Ensembl 2024. Nucleic Acids Res. 2024;52(D1):D891–D899.
Disponible en: https://doi.org/10.1093/nar/gkad1049
Contreras-Castillo S, Plaza A, Stojanova J, Navarro G, Carmona R,
Corvalán F, et al. Effect of CYP3A4, CYP3A5, MDR1 and POR genetic
polymorphisms in immunosuppressive treatment in Chilean kidney
transplanted patients. Front Pharmacol. 2021;12:674117. Disponible
en: https://doi.org/10.3389/fphar.2021.674117
Zaruma-Torres F, Lares-Asseff I, Lima A, Reyes-Espinoza A, Loera-
Castañeda V, Sosa-Macías M, et al. Genetic polymorphisms
associated to folate transport as predictors of increased risk for
acute lymphoblastic leukemia in Mexican children. Front Pharmacol.
;7:238. Disponible en: https://doi.org/10.3389/fphar.2016.00238
Pagnotta PA, Melito VA, Lavandera JV, Parera VE, Rossetti MV,
Zuccoli JR, et al. Role of ABCB1 and glutathione S-transferase gene
variants in the association of porphyria cutanea tarda and human
immunodeficiency virus infection. Biomed Rep. 2021;14(2):22.
Disponible en: https://doi.org/10.3892/br.2020.1398
De Castro ANCL, Fernandes MR, de Carvalho DC, de Souza TP, Rodrigues
JCG, Andrade RB, et al. Polymorphisms of xenobiotic-metabolizing
and transporter genes, and the risk of gastric and colorectal cancer
in an admixed population from the Brazilian Amazon. Am J Transl
Res. 2020;12(10):6626-36. Disponible en: https://doi.org/10.21203/
rs.2.14223/v1
Ruiz-Linares A, Adhikari K, Acuña-Alonzo V, et al. Admixture in
Latin America: geographic structure, phenotypic diversity and selfperception
of ancestry based on 7,342 individuals. PLoS Genet.
;10(9):e1004572. Disponible en: https://doi.org/10.1371/
journal.pgen.1004572
Sandoval JR, Lacerda DR, Acosta O, Jota MS, Robles-Ruiz P, Salazar-
Granara A, et al. The Genetic History of Peruvian Quechua-Lamistas
and Chankas: Uniparental DNA Patterns among Autochthonous
Amazonian and Andean Populations. Ann Hum Genet. 2016;80(2):88-
Disponible en: https://doi.org/10.1111/ahg.12145
Fitz-James MH, Cavalli G. Molecular mechanisms of transgenerational
epigenetic inheritance. Nat Rev Genet. 2022;23(6):325-341.
Disponible en: https://doi.org/10.1038/s41576-021-00438-5
Bigham AW. Genetics of human origin and evolution: high-altitude
adaptations. Curr Opin Genet Dev. 2016;41:8-13. Disponible en:
https://doi.org/10.1016/j.gde.2016.06.018
Song W, Shi Y, Wang W, Pan W, Qian W, Yu S, et al. A selection pressure
landscape for 870 human polygenic traits. Nat Hum Behav.
;5(12):1731-1743. Disponible en: https://doi.org/10.1038/
s41562-021-01231-4
Pena SDJ, Santos FR, Tarazona‐Santos, E. Genetic admixture in Brazil.
Am J Med Genet C Semin Med Genet. 2020;184(4):928-938. Disponible
en: https://doi.org/10.1002/ajmg.c.31853
Han E, Carbonetto P, Curtis RE, Wang Y, Granka JM, Byrnes J, et
al. Clustering of 770,000 genomes reveals post-colonial population
structure of North America. Nat Commun. 2017;8:14238. Disponible
en: https://doi.org/10.1038/ncomms14238
Bryc K, Durand EY, Macpherson JM, Reich D, Mountain JL. The genetic
ancestry of African Americans, Latinos, and European Americans
across the United States. Am J Hum Genet. 2015;96(1):37-53.
Disponible en: https://doi.org/10.1016/j.ajhg.2014.11.010
González-Medina L, Barquera R, Delgado-Aguirre H, Clayton S, Adalid-
Sáinz C, Arrieta-Bolaños E, et al. Genetic diversity of HLA system
in two populations from Durango, Mexico: Durango city and rural
Durango. Hum Immunol. 2020;81(9):489-491. Disponible en: https://
doi.org/10.1016/j.humimm.2019.06.005
Ziyatdinov A, Torres J, Alegre-Díaz J, Backman J, Mbatchou J, Turner
M, et al. Genotyping, sequencing and analysis of 140,000 adults from
Mexico City. Nature. 2023;622(7984):784–793. Disponible en: https://
doi.org/10.1038/s41586-023-06595-3
Whirl-Carrillo M, Huddart R, Gong L, Sangkuhl K, Thorn CF,
Whaley R, et al. An evidence-based framework for evaluating
pharmacogenomics knowledge for personalized Medicine. Clin
Pharmacol Ther. 2021;110(3):563-572. Disponible en: https://doi.
org/10.1002/cpt.2350
Jin H, Zhang C, Zwahlen M, von Feilitzen K, Karlsson M, Shi M, et
al. Systematic transcriptional analysis of human cell lines for gene
expression landscape and tumor representation. Nat Commun. 2023
(1):5417. Disponible en: https://doi.org/10.1038/s41467-023-
-w
Uhlen M, Zhang C, Lee S, Sjöstedt E, Fagerberg L, Bidkhori G, et
al. A pathology atlas of the human cancer transcriptome. Science.
;357(6352):eaan2507. Disponible en: https://doi.org/10.1126/
science.aan2507
Karlsson M, Zhang C, Méar L, Zhong W, Digre A, Katona B, et
al. A single-cell type transcriptomics map of human tissues. Sci
Adv. 2021;7(31):eabh2169. Disponible en: https://doi.org/10.1126/
sciadv.abh2169
Uhlén M, Fagerberg L, Hallström BM, Lindskog C, Oksvold P, Mardinoglu
A, et al. Proteomics: tissue-based map of the human proteome.
Science. 2015;347(6220):1260419. Disponible en: https://doi.
org/10.1126/science.1260419
Downloads
Publicado
Como Citar
Edição
Seção
Licença
Copyright (c) 2025 Horizonte Médico (Lima)

Este trabalho está licenciado sob uma licença Creative Commons Attribution 4.0 International License.
Os resultados de pesquisa da revista Horizonte Médico (Lima) (Horiz. Med.) são publicados sem custo e estão disponíveis gratuitamente para download sob o modelo de acesso aberto, com o objetivo de disseminar trabalhos e experiências desenvolvidos nas áreas biomédica e de saúde pública, tanto nacional quanto internacionalmente, e promover a pesquisa nos diferentes campos da medicina humana. Todos os manuscritos aceitos e publicados na revista são distribuídos gratuitamente sob os termos de uma licença Creative Commons - Atribuição 4.0 Internacional (CC BY 4.0).

















